NumPy 实现k均值聚类算法(k-means)
机器学习算法与Python实践这个系列主要是参考《机器学习实战》这本书。因为自己想学习Python,然后也想对一些机器学习算法加深下了解,所以就想通过Python来实现几个比较常用的机器学习算法。恰好遇见这本同样定位的书籍,所以就参考这本书的过程来学习了。
机器学习中有两类的大问题,一个是分类,一个是聚类。分类是根据一些给定的已知类别标号的样本,训练某种学习机器,使它能够对未知类别的样本进行分类。这属于supervised learning(监督学习)。而聚类指事先并不知道任何样本的类别标号,希望通过某种算法来把一组未知类别的样本划分成若干类别,这在机器学习中被称作 unsupervised learning (无监督学习)。在本文中,我们关注其中一个比较简单的聚类算法:k-means算法。
k-means算法简介
通常,人们根据样本间的某种距离或者相似性来定义聚类,即把相似的(或距离近的)样本聚为同一类,而把不相似的(或距离远的)样本归在其他类。
我们以一个二维的例子来说明下聚类的目的。如下图左所示,假设我们的n个样本点分布在图中所示的二维空间。从数据点的大致形状可以看出它们大致聚为三个cluster,其中两个紧凑一些,剩下那个松散一些。我们的目的是为这些数据分组,以便能区分出属于不同的簇的数据,如果按照分组给它们标上不同的颜色,就是像下图右边的图那样:
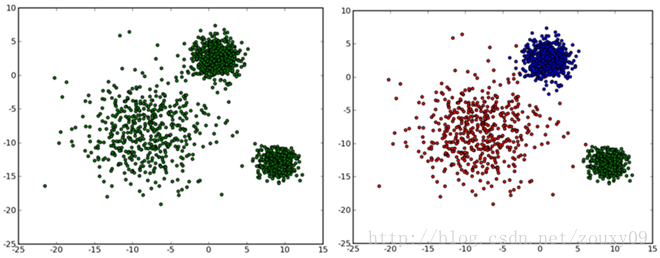
如果人可以看到像上图那样的数据分布,就可以轻松进行聚类。但我们怎么教会计算机按照我们的思维去做同样的事情呢?这里就介绍个集简单和经典于一身的k-means算法。
k-means算法是一种很常见的聚类算法,它的基本思想是:通过迭代寻找k个聚类的一种划分方案,使得用这k个聚类的均值来代表相应各类样本时所得的总体误差最小。
k-means算法的基础是最小误差平方和准则。其代价函数是:
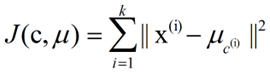
上式中,μc(i)表示第i个聚类的均值。我们希望代价函数最小,直观的来说,各类内的样本越相似,其与该类均值间的误差平方越小,对所有类所得到的误差平方求和,即可验证分为k类时,各聚类是否是最优的。
上式的代价函数无法用解析的方法最小化,只能有迭代的方法。k-means算法是将样本聚类成 k个簇(cluster),其中k是用户给定的,其求解过程非常直观简单,具体算法描述如下:
- 随机选取 k个聚类质心点
- 重复下面过程直到收敛
- 对于每一个样例 i,计算其应该属于的类:
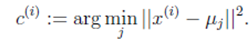
- 对于每一个类 j,重新计算该类的质心:
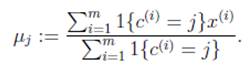
- 对于每一个样例 i,计算其应该属于的类:
下图展示了对n个样本点进行K-means聚类的效果,这里k取2。
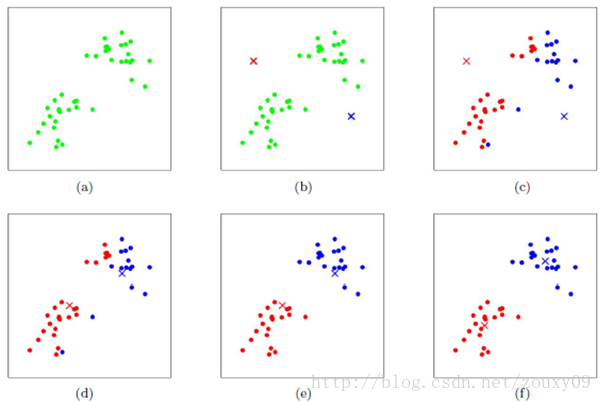
其伪代码如下:
创建k个点作为初始的质心点(随机选择)
当任意一个点的簇分配结果发生改变时
对数据集中的每一个数据点
对每一个质心
计算质心与数据点的距离
将数据点分配到距离最近的簇
对每一个簇,计算簇中所有点的均值,并将均值作为质心
Numpy 实现
我使用的Python是2.7.5版本的。附加的库有Numpy和Matplotlib。具体的安装和配置见前面的博文。在代码中已经有了比较详细的注释了。不知道有没有错误的地方,如果有,还望大家指正(每次的运行结果都有可能不同)。里面我写了个可视化结果的函数,但只能在二维的数据上面使用。直接贴代码:
kmeans.py:
#################################################
# kmeans: k-means cluster
# Author : zouxy
# Date : 2013-12-25
# HomePage : http://blog.csdn.net/zouxy09
# Email : zouxy09@qq.com
#################################################
from numpy import *
import time
import matplotlib.pyplot as plt
# calculate Euclidean distance
def euclDistance(vector1, vector2):
return sqrt(sum(power(vector2 - vector1, 2)))
# init centroids with random samples
def initCentroids(dataSet, k):
numSamples, dim = dataSet.shape
centroids = zeros((k, dim))
for i in range(k):
index = int(random.uniform(0, numSamples))
centroids[i, :] = dataSet[index, :]
return centroids
# k-means cluster
def kmeans(dataSet, k):
numSamples = dataSet.shape[0]
# first column stores which cluster this sample belongs to,
# second column stores the error between this sample and its centroid
clusterAssment = mat(zeros((numSamples, 2)))
clusterChanged = True
## step 1: init centroids
centroids = initCentroids(dataSet, k)
while clusterChanged:
clusterChanged = False
## for each sample
for i in xrange(numSamples):
minDist = 100000.0
minIndex = 0
## for each centroid
## step 2: find the centroid who is closest
for j in range(k):
distance = euclDistance(centroids[j, :], dataSet[i, :])
if distance < minDist:
minDist = distance
minIndex = j
## step 3: update its cluster
if clusterAssment[i, 0] != minIndex:
clusterChanged = True
clusterAssment[i, :] = minIndex, minDist**2
## step 4: update centroids
for j in range(k):
pointsInCluster = dataSet[nonzero(clusterAssment[:, 0].A == j)[0]]
centroids[j, :] = mean(pointsInCluster, axis = 0)
print 'Congratulations, cluster complete!'
return centroids, clusterAssment
# show your cluster only available with 2-D data
def showCluster(dataSet, k, centroids, clusterAssment):
numSamples, dim = dataSet.shape
if dim != 2:
print "Sorry! I can not draw because the dimension of your data is not 2!"
return 1
mark = ['or', 'ob', 'og', 'ok', '^r', '+r', 'sr', 'dr', '<r', 'pr']
if k > len(mark):
print "Sorry! Your k is too large! please contact Zouxy"
return 1
# draw all samples
for i in xrange(numSamples):
markIndex = int(clusterAssment[i, 0])
plt.plot(dataSet[i, 0], dataSet[i, 1], mark[markIndex])
mark = ['Dr', 'Db', 'Dg', 'Dk', '^b', '+b', 'sb', 'db', '<b', 'pb']
# draw the centroids
for i in range(k):
plt.plot(centroids[i, 0], centroids[i, 1], mark[i], markersize = 12)
plt.show()
测试
测试数据是二维的,共80个样本。有4个类。如下:
testSet.txt:
测试结果:
1.658985 4.285136
-3.453687 3.424321
4.838138 -1.151539
-5.379713 -3.362104
0.972564 2.924086
-3.567919 1.531611
0.450614 -3.302219
-3.487105 -1.724432
2.668759 1.594842
-3.156485 3.191137
3.165506 -3.999838
-2.786837 -3.099354
4.208187 2.984927
-2.123337 2.943366
0.704199 -0.479481
-0.392370 -3.963704
2.831667 1.574018
-0.790153 3.343144
2.943496 -3.357075
-3.195883 -2.283926
2.336445 2.875106
-1.786345 2.554248
2.190101 -1.906020
-3.403367 -2.778288
1.778124 3.880832
-1.688346 2.230267
2.592976 -2.054368
-4.007257 -3.207066
2.257734 3.387564
-2.679011 0.785119
0.939512 -4.023563
-3.674424 -2.261084
2.046259 2.735279
-3.189470 1.780269
4.372646 -0.822248
-2.579316 -3.497576
1.889034 5.190400
-0.798747 2.185588
2.836520 -2.658556
-3.837877 -3.253815
2.096701 3.886007
-2.709034 2.923887
3.367037 -3.184789
-2.121479 -4.232586
2.329546 3.179764
-3.284816 3.273099
3.091414 -3.815232
-3.762093 -2.432191
3.542056 2.778832
-1.736822 4.241041
2.127073 -2.983680
-4.323818 -3.938116
3.792121 5.135768
-4.786473 3.358547
2.624081 -3.260715
-4.009299 -2.978115
2.493525 1.963710
-2.513661 2.642162
1.864375 -3.176309
-3.171184 -3.572452
2.894220 2.489128
-2.562539 2.884438
3.491078 -3.947487
-2.565729 -2.012114
3.332948 3.983102
-1.616805 3.573188
2.280615 -2.559444
-2.651229 -3.103198
2.321395 3.154987
-1.685703 2.939697
3.031012 -3.620252
-4.599622 -2.185829
4.196223 1.126677
-2.133863 3.093686
4.668892 -2.562705
-2.793241 -2.149706
2.884105 3.043438
-2.967647 2.848696
4.479332 -1.764772
-4.905566 -2.911070
test_kmeans.py
测试代码:
#################################################
# kmeans: k-means cluster
# Author : zouxy
# Date : 2013-12-25
# HomePage : http://blog.csdn.net/zouxy09
# Email : zouxy09@qq.com
#################################################
from numpy import *
import time
import matplotlib.pyplot as plt
## step 1: load data
print "step 1: load data..."
dataSet = []
fileIn = open('E:/Python/Machine Learning in Action/testSet.txt')
for line in fileIn.readlines():
lineArr = line.strip().split('\t')
dataSet.append([float(lineArr[0]), float(lineArr[1])])
## step 2: clustering...
print "step 2: clustering..."
dataSet = mat(dataSet)
k = 4
centroids, clusterAssment = kmeans(dataSet, k)
## step 3: show the result
print "step 3: show the result..."
showCluster(dataSet, k, centroids, clusterAssment)
运行的前后结果对比:
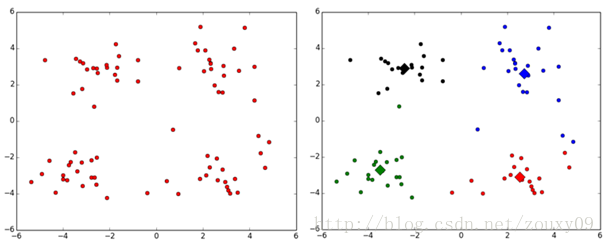
不同的类用不同的颜色来表示,其中的大菱形是对应类的均值质心点。
算法分析
k-means算法比较简单,但也有几个比较大的缺点:
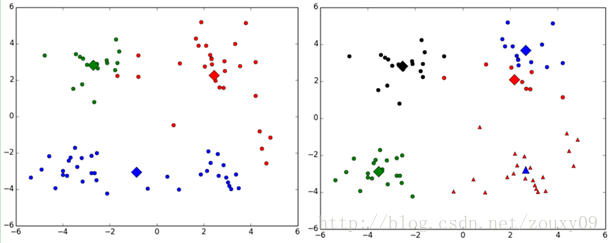
- (1)k值的选择是用户指定的,不同的k得到的结果会有挺大的不同,如下图所示,左边是k=3的结果,这个就太稀疏了,蓝色的那个簇其实是可以再划分成两个簇的。而右图是k=5的结果,可以看到红色菱形和蓝色菱形这两个簇应该是可以合并成一个簇的:
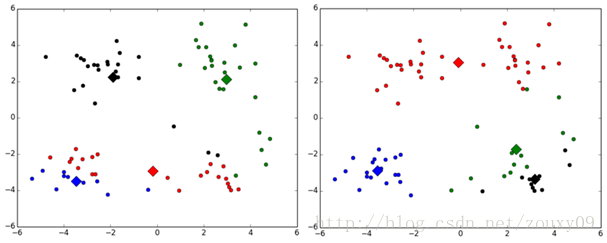
- (2)对k个初始质心的选择比较敏感,容易陷入局部最小值。例如,我们上面的算法运行的时候,有可能会得到不同的结果,如下面这两种情况。K-means也是收敛了,只是收敛到了局部最小值:
- (3)存在局限性,如下面这种非球状的数据分布就搞不定了:
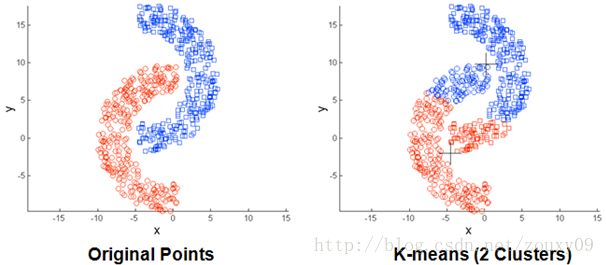
- (4)数据库比较大的时候,收敛会比较慢。
k-means老早就出现在江湖了。所以以上的这些不足也被世人的目光敏锐的捕捉到,并融入世人的智慧进行了某种程度上的改良。例如问题(1)对k的选择可以先用一些算法分析数据的分布,如重心和密度等,然后选择合适的k。而对问题(2),有人提出了另一个成为二分k均值(bisecting k-means)算法,它对初始的k个质心的选择就不太敏感,这个算法我们下一个博文再分析和实现。
文章出处
作者:zouxy09 来源:CSDN 原文:https://blog.csdn.net/zouxy09/article/details/17589329